VERONIKA MUELLER AND CHRISTIAN EGGELING, MAX PLANCK INSTITUTE; HÅKAN KARLSSON, COBOLT AB; AND DAG VON GEGERFELT, VON GEGERFELT PHOTONICS
到目前为止,实验室中的受激发射损耗(STED)显微镜通常使用大而复杂的激光系统。但是使用紧凑、低噪音、0.5W、660 nm、单频连续二极管泵浦固态(DPSS)激光器来搭建STED显微镜系统可以明显的降低系统的额尺寸复杂度与花费,与此同时还可以提供和标准的STED显微镜同样的精度。
按照阿贝定律(18世纪70年代,德国物理学家恩斯特·阿贝发现,可见光由于其波动特性会发生衍射,因而光束不能无限聚焦。根据这个阿贝定律,可见光能聚焦的最小直径是光波波长的三分之一,也就是200纳米。一个多世纪以来,200纳米的“阿贝极限”一直被认为是光学显微镜理论上的分辨率极限,小于这个尺寸的物体必须借助电子显微镜或隧道扫描显微镜才能观察。)的描叙,远场荧光显微镜的分辨率由于光的衍射存在一个极限。以标准的共焦显微镜为例,可见激发光束的最小分辨率为200nm。激光束在样品上扫描得到的图像的细节在比这个极限小的尺寸下产生了模糊,如图1a所示。
近几十年来,这种物理上的限制对进一步了解细胞结构和细胞功能造成了阻碍,很多重要的细节也因此模糊不清。而Stefan Hell创造性的发明了选择性地开关荧光这种方法来突破这个屏障,带来了具有纳米分辨尺度的荧光显微镜。这些纳米显微技术都是基于通过基于可逆开关的荧光激发。
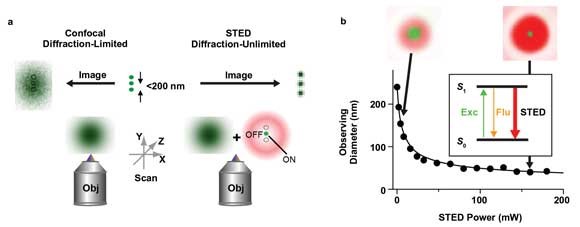
图1. 原理远场扫描STED纳米显微镜的。(1)当存在着衍射极限(大于200nm)的传统共焦显微镜不能区分距离小于200nm的物体,会导致成像模糊,而STED纳米分辨技术可以通过开关荧光来得到超高的分辨率,其中每次观测时只有几个荧光团发光,其他的荧光团都处于湮灭状态,Obj为物镜。(2)STED纳米显微镜的分辨率随着STED激光强度的增加而增加:通过用STED激光光束覆盖激发光的衍射极限,这样形成了一个区域荧光强度为0(用红色表示),而这个区域的中心依然存在荧光(用绿色标识,即观测区域)。插图:被激发到激发态的荧光(flu,橙色)团又被STED湮灭到了基态。
在这种受激发射损耗(STED)显微镜中,激发光束被红移的激光光束覆盖,红移的激光光束具有一个或者更多个0强度点。当红移的STED光束的强度到达某个阈值时,非0强度区域的荧光就被选择性的湮灭了,而0强度点—它的大小远小于衍射极限200 nm—内的荧光团将会继续发出荧光。具体原理如图1所示。而观测体积的减小受到STED光束强度影响,所以该技术的瓶颈在于技术而不是原理。这种技术得到的荧光图像在原理上没有空间分辨率的上限。科研工作者已经利用该技术对20 nm精度的细胞内分子分布进行了高速成像,这能够显著的推动分子生物学研究的前进。STED技术使得像细胞膜异质性工作原理、组织或者体内的神经元细胞的活性依赖这样在以往很难进行的研究得以实现。
STED显微镜原型的缺点是相对来说较高的复杂度以及对于大而昂贵的高功率脉冲激光器的需求,这样的激光器包括钛蓝宝石泵浦超快光参量振荡器。但是随着新荧光团的发现以及激光技术的发展,STED显微镜的实用性,特别是在活细胞内成像领域,有了显著的改善。比如,你可以选择合适的激光波长来用STED纳米显微镜对在活细胞内的绿色荧光蛋白进行成像。
活细胞STED稳步增加
当扫描速度能达到几千赫兹时,活细胞STED技术就能更进一步的发展。因为这样的扫描速度就避免了荧光团在三重态或者其他的(“成像不稳定”)暗态的堆积。这样的扫描速度加上连续STED激光以及门控检测方案,就能够显著的减少光损伤。此外,最近在荧光标记蛋白上的进展,比如SNAP标记,可以做到光稳定同时把有机染料运输到活细胞内(图2a)。
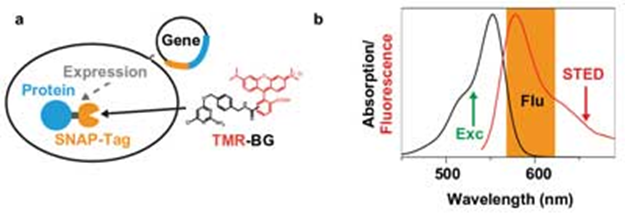
图2.四甲基若丹明(TMR)SNAP标记,(a)通过融合蛋白把感兴趣的蛋白(蓝色)和SNAP标记(黄色)融合到一起来实现用TMR标记活细胞内的特定蛋白。在培育过程中,官能染料TMR-BG(BG=苄基)进入细胞内,共价结合到SNAP标记。(b)激发波长为532 nm(绿色)TMR在水中的的吸收(黑色)和荧光(红色)光谱,黄色的是荧光探测窗口,优化的STED波长为660 nm。
现在,我们在世界上首次用紧凑型低噪音的CW二极管泵浦固态(DPSS)红色激光集成到了STED显微镜之中。使用这样的激光器的大大降低了纳米显微镜装置的复杂度、尺寸和成本。试验中用的染料是细胞膜可渗透的罗丹明(四甲基)TMR染料,这是STED纳米显微镜SNAP标记技术非常有用的染料。这种染料需要的STED波长在650~670 nm范围内(图2b)。
激光用的是瑞典Cobolt AB公司的660 nm DPSS型激光器,它基于小型化的环形谐振腔设计,集成到一个热控和密封包装。它具有稳定的功率输出和低强度的噪声,可以用在TMR
标记的细胞样品纳米显微镜上;这些限制还使其支持STED和以单分子为基础的光谱手段相结合。比如用荧光相关光谱(FCS)结合STED研究脂质和蛋白质的纳米膜扩散。
用脉冲激发和门控探测结合连续激光器用在STED显微镜上,不仅仅用CW-STED方式可以消除了模糊和固有的对比度降低,还可以用打到样品上的激光功率为100~150 mW的CW-STED激光来进行活细胞成像。
用门控技术记录TMR-SNAP标记的STED图像这项技术对于CW-STED激光的性能提出了相当高的要求。除了上文提及的波长在650~670 nm范围内,同时还需要打到样品上的激光功率在100~150 mW之间,考虑到纳米显微镜光学器件之间的损耗,我们需要红色激光器的功率至少为400 mW。
另一个重要的先决条件是激光器的噪声很低,因为在功率上任何波动都会反映到最终图像的对比度。图3就是用门控技术结合低噪音的DPSS激光器得到的STED图像,使用的激光器是Cobolt Flamenco,它的功率和波长分别是500 mW和660 nm,噪声的均方根值小于0.1%。
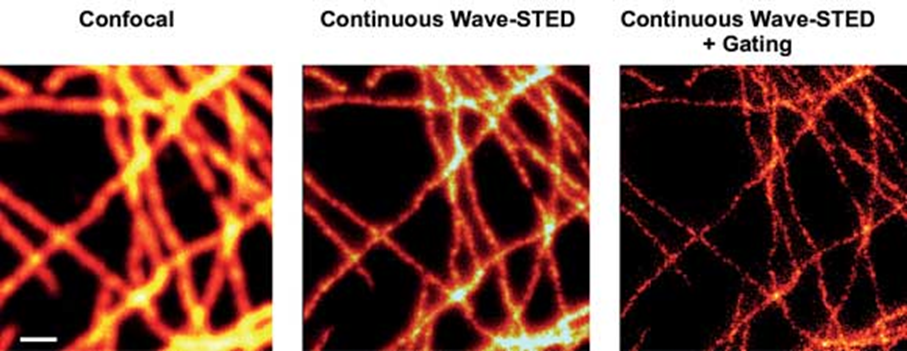
图3,用波长660 nm二极管泵浦固体激光器的TMR标记细胞的STED图像。从左到右分别是扫描共焦,CW-STED和门控+CW-STED得到的图像。观测的对象是哺乳动物细胞的微管。比例尺:500纳米。
出色的分辨率
样品功率为130 mW时得到的STED图像显示出了比有衍射限制的共焦图像高得多的分辨率,实验用来对比的对象是哺乳动物细胞的微管,技术是SNAP标记,标记物是TMR。激发激光是用的Picoquant GmbH的532 nm激光。因此,使用一个小的CW DPSS激光器显著的降低了STED纳米显微镜设置的成本和复杂度,而不会损害其性能。
细胞信号研究领域一个重要的主题是细胞质膜上多样组织的角色和分子动力学。众所周知,脂质-脂质和脂质-蛋白质相互作用所带来的细胞膜异质性参与了细胞膜膜相关过程。这些反应的空间尺度通常都小于200 nm。因此用常规的远场显微镜直接和非侵入性的观察在活细胞内的这些反应都受到了分辨率极限的限制。
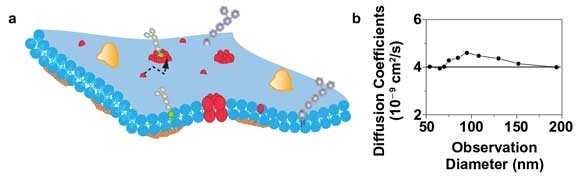
图4.用660 nm纳米二极管泵浦固体激光器得到了细胞膜动力学STED-FCS结果。(a)细胞质膜的机理图示,其中脂质会和其他分子的相互作用导致的扩散(虚线)不均匀;(b)活哺乳动物细胞的细胞质膜中用TMR标记的磷酸乙醇胺脂质的扩散系数,扩散系数的确定是来自于不同的FCS尺寸得到的数据。横线是根据自由布朗扩散推算出的0.4μm
2/s的扩散系数常数。
STED和FCS的结合
由于远场STED显微镜所具有的超高的分辨率,它可以直接用于研究细胞膜的不均匀性。比如,除了提供对蛋白质纳米簇成像所需要的分辨率之外,STED纳米显微镜还是和FCS相结合来研究纳米尺度的复杂动力过程。在FCS的研究中,我们把随着时间的荧光抖动当作标记分子在测量范围内的出入。由于轻微的障碍扩散,比如异质性和分子间的相互作用,导致在观测区域的传输时间变化,STED-FCS能够观察这些分子在纳米级尺度上的相互作用。在之前的STED-FCS研究中,科研工作者发现这项技术可以用来研究荧光标记的脂质分子的自由与受阻运动之间的差异。某些鞘脂类似物瞬间(约10毫秒)被胆固醇介导分子复合物捕获,而其他一些化合物,比如磷酸甘油脂质类似物,就是自由扩散的。
门控技术以及CW-STED激光器由于上文提及的原因也促进了STED-FCS及成像技术的发展。由于噪音意味着荧光信号的抖动,STED-FCS技术对于激光器的低噪音有特殊的需求,如果噪声水平得到改进的话将会显著的改善抖动的幅度。因此我们在活哺乳动物细胞的质膜中TMR标记的磷脂的STED-FCS试验中再次采用了660 nm低噪音的DPSS激光。
图4b是对于不同观测尺寸(可以通过增加STED的功率和调整门控的时间来实现)的STED-FCS研究得到的扩散系数。这个实验中,对于脂质扩散的准确观察可以精确到50 nm的直径下。从运输时间和相应的被观察区域的大小可以计算出不同记录过程的脂质表观扩散系数。从实验结果可以看出,表观扩散系数相对于研究区域尺寸独立,即这些磷脂分子和我们估计的一样是在做自由布朗运动。532 nm激发和660 nm的STED也让STED-FCS技术使用TMR-SNAP标记成为了现实。这是细胞膜异质性的一个特殊的要求,因为SNAP标记可以标记细胞内外的(跨)膜蛋白,这样就可以最小化标记对于它们功能的改变。
References
1. S.W. Hell and J. Wichmann (1994). Breaking the diffraction resolution limit by stimulated-emission - stimulated-emission-depletion fluorescence microscopy. Opt Lett, Vol. 19, pp. 780-782.
2. S.W. Hell (2009). Microscopy and its focal switch. Nat Methods, Vol. 6, pp. 24-32.
3. G. Donnert et al (2006). Macromolecular-scale resolution in biological fluorescence microscopy. PNAS, Vol. 103, pp. 11440-11445.
4. J.J. Sieber et al (2007). Anatomy and dynamics of a supramolecular membrane protein cluster. Science, Vol. 317, pp. 1072-1076.
5. C. Eggeling et al (2009). Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature, Vol. 457, pp. 1159-U1121.
6. U.V. Nagerl et al (2008). Live-cell imaging of dendritic spines by STED microscopy. PNAS, Vol. 105, pp. 18982-18987.
7. S. Berning et al (2012). Nanoscopy in a living mouse brain. Science, Vol. 335, p. 551.
8. B.R. Rankin et al (2011). Nanoscopy in a living multicellular organism expressing GFP. Biophys J, Vol. 100, pp. L63–L65.
9. G. Donnert et al (2007). Major signal increase in fluorescence microscopy through dark-state relaxation. Nat Methods, Vol. 4, pp. 81-86.
10. G. Moneron et al (2010). Fast STED microscopy with continuous wave fiber lasers. Opt Express, Vol. 18, pp. 1302-1309.
11. A. Keppler et al (2003). A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat Biotechnol, Vol. 21, pp. 86-89.
12. B. Hein et al (2010). Stimulated emission depletion nanoscopy of living cells using SNAP-tag fusion proteins. Biophys J, Vol. 98, pp. 158-163.
13. J.R. Moffitt et al (2011). Time-gating improves the spatial resolution of STED microscopy. Opt Express, Vol. 19, pp. 4242-4254.
14. G. Vicidomini et al (2011). Sharper low-power STED nanoscopy by time gating. Nat Methods, Vol. 8, pp. 571-573.
15. D. Lingwood and K. Simons (2010). Lipid rafts as a membrane-organizing principle. Science, Vol. 327, pp. 46-50.
16. C. Ringemann et al (2009). Exploring single-molecule dynamics with fluorescence nanoscopy. New J Phys, Vol. 11, p. 103054.
17. V. Mueller et al (2011). STED nanoscopy reveals molecular details of cholesterol- and cytoskeleton-modulated lipid interactions in living cells. Biophys J, Vol. 101, pp. 1651-1660.
(翻译:北京鼎信优威光子科技有限公司 汪 阳博士)



